AMBERの原子選択(アトムマスク)を完全に理解する
本記事はAMBER-Hub Atom Mask Selection Syntaxの内容を基に作成しています。
1. 基本
cpptrajの原子指定では、ある特別な文字(:, @, *) の後に、残基番号や原子名等を指定することで原子を選択することができます。
2. 残基・原子・チェーンの選択
2.1 残基の選択
残基は、:に続いて残基番号や残基名を指定することで選択することができます。
残基番号を指定する場合、-を用いることで範囲を、,を用いることで複数範囲を指定することができます。
# 基本構文
:(残基番号または残基名)
# 残基番号2番を選択
:2
# 残基番号1から10番までを選択
:1-10
# 残基番号1から10番までと、15番を選択
:1-10,15
# 全てのアラニンを選択
:ALA
# 全てのアラニンとリシンを選択
:ALA,LYS
:2残基番号2番
:2,5-7残基番号2番と5から7番
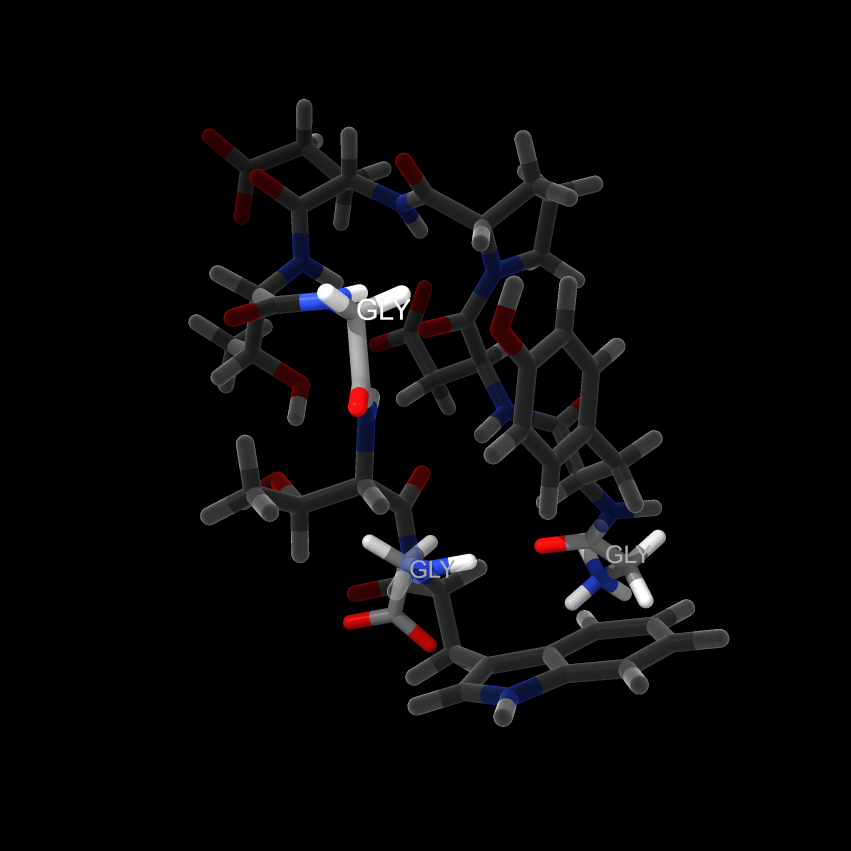
:GLY全てのグリシン残基
2.2 原子の選択
原子は@に続いてファイル中の原子番号や原子要素名を指定することで選択することができます。
残基の場合と同様に-を用いることで範囲を、,を用いることで複数範囲を指定することができます。
@に続いて\や%を記述することで、それぞれ元素名と原子タイプを指定することができます。
原子の要素名で選択する場合、必ずしも元素記号と一致しないことに注意が必要です。例えば、酸素を指定したい場合、@Oだと全ての酸素を指定できない場合が多いです。あくまで、“O”という原子要素名がついた原子のみが選択されます。すべての酸素を選択したい場合は、原子要素名を指定する@ではなく、元素を指定する@/を使うか、第3章で解説するワイルドカードを使います。
# 基本構文
@(残基番号または残基名)
@\(元素名)
@%(原子タイプ名)
# ファイル中の1番原子を選択
@1
# 1番原子から10番原子までを選択
@1-10
# 1番原子から10番原子までと、15番原子を選択
@1-10,15
# 全てのCα原子を選択
@CA
# 全ての原子要素名"O"と"N"を選択
@O,N
# 全ての酸素原子と窒素を選択
@\O,N
# 全ての原子タイプCTを選択
@%CT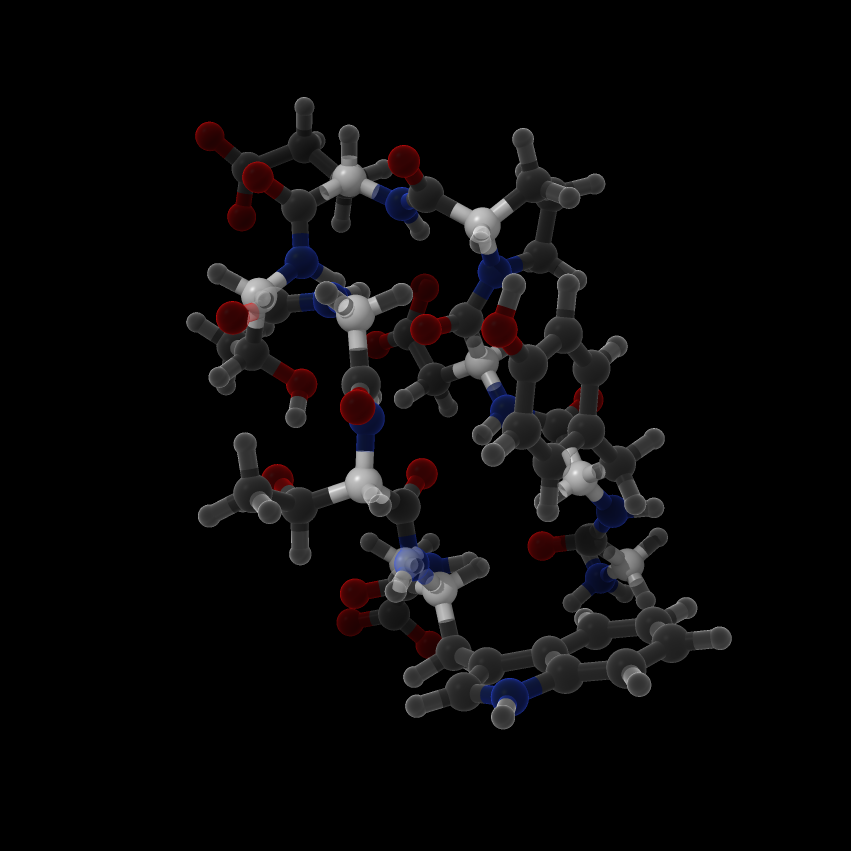
@CA原子要素名CA(タンパク質のCα原子)
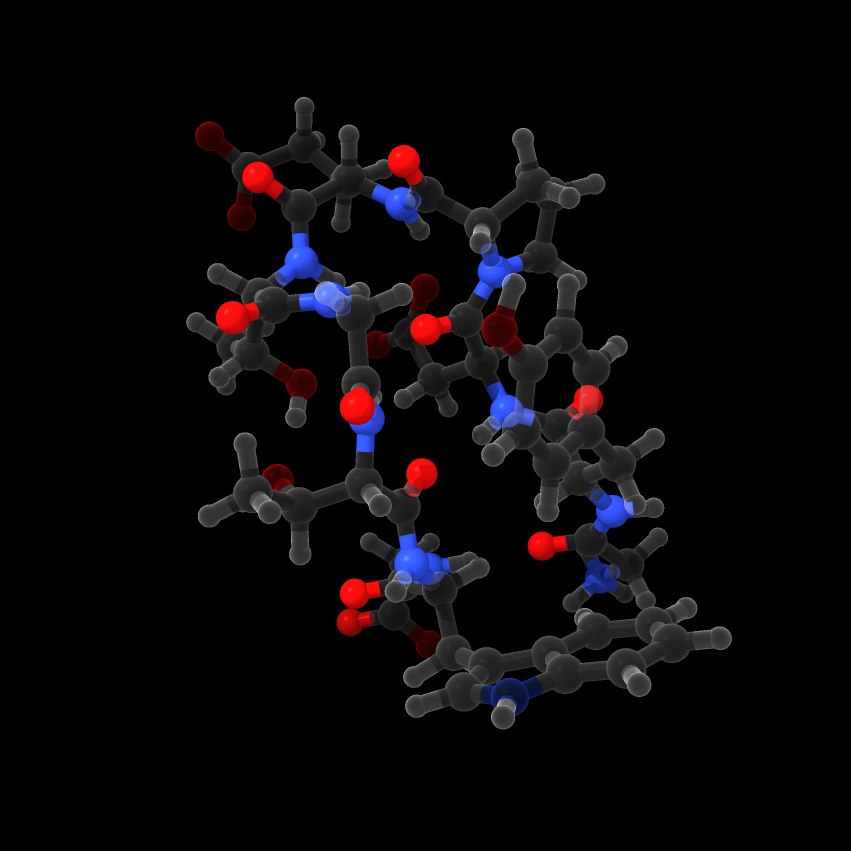
@O,N原子要素名OとN
この選択だと原子要素名が”O”と”N”の原子のみが選択される。酸素原子であっても、OG1,OD1,OHなど、ほかの原子要素名は選択されない。

@/O,N酸素原子と窒素原子
これで全ての酸素原子と窒素原子が選択される。
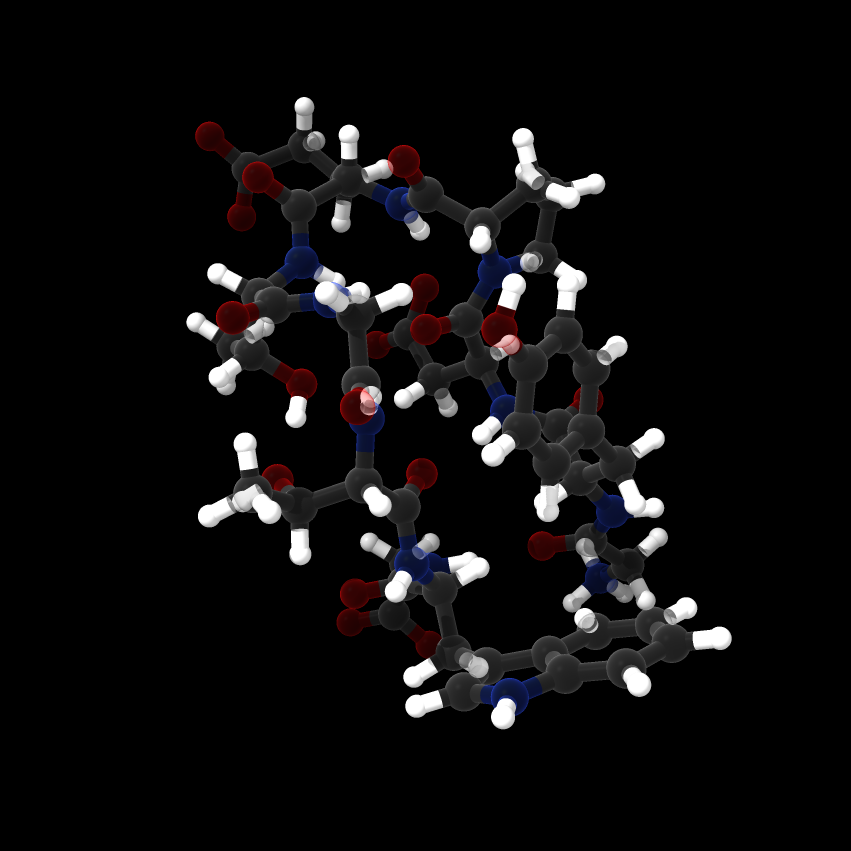
@H*全ての水素原子
第3章で解説するワイルドカードを用いても元素での選択ができる。
2.3 チェーン(鎖)の選択
PDBファイルによってはファイル内に複数のチェーンが含まれている場合があります。
チェーンは::に続いてチェーンIDを指定することで選択できます。
# 基本構文
::(チェーンID)
# A鎖を選択
::A
# A鎖とC鎖を選択
::A,C
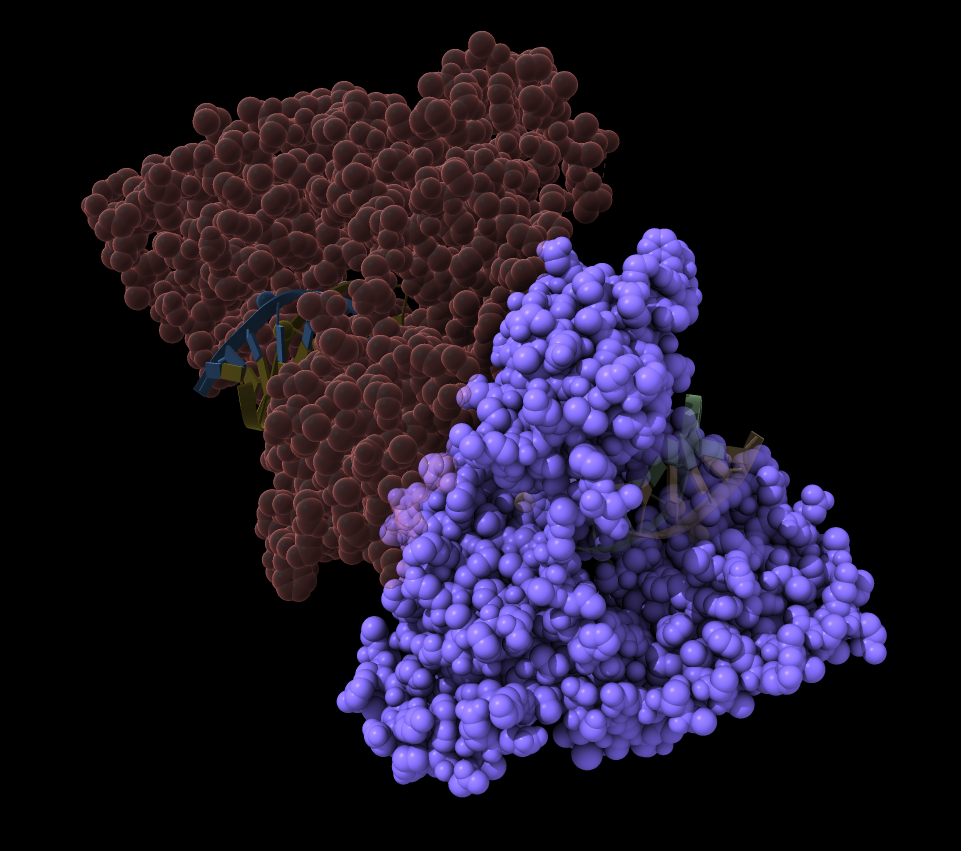
::AチェーンA
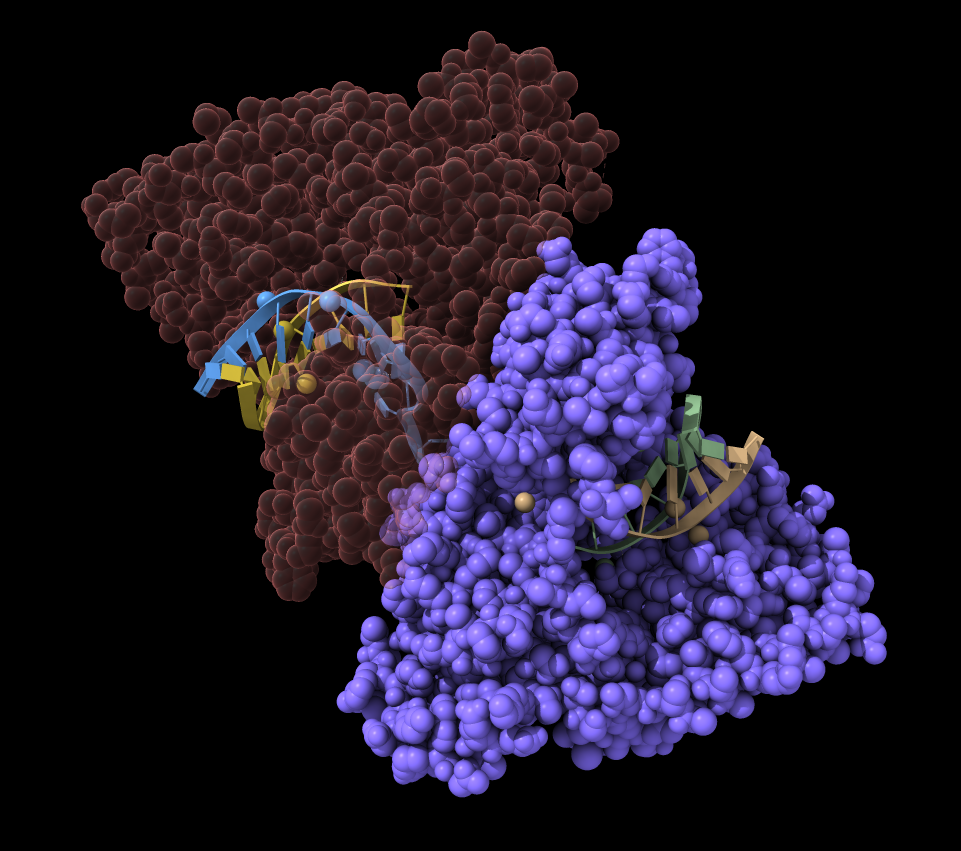
::A,C,P,D,TチェーンA,C,P,D,T
2.4 チェーンと残基と原子を組み合わせて指定する
これらはすべて組み合わせて選択することができます。
組み合わせる場合、基本的にチェーン、残基、原子の順番で指定します。
例えば、A鎖のリシンのCα原子を選択したい場合、次のように記述します。
# A鎖のリシンのCα原子
::A:LYS@CA3. ワイルドカードを使う
cpptrajの原子選択ではワイルドカードを用いることができます。 ワイルドカードとは、任意の文字列のことです。これを使うことで、例えばHで始まる原子要素名を一気に指定することができます。
cpptrajで使うことができるワイルドカード文字は*, =, ?の3つです。
*: 0文字以上の任意の文字列(0文字でも大丈夫)=: 0文字以上の任意の文字列(*と同じ意味)?: 1文字の任意の文字列
使用例
# 原子要素名がHで始まるすべての原子(全水素原子)(@H,H1,H2,H......)
@H*
@H=
# 残基番号の下一桁が'5'で二桁番目に存在する残基 (:15,25,35,45,55,65,75,85,95)
:?54. 論理演算子を使う
論理演算子も用いることができます。AND, OR, NOTにあたる&, |, !の3つがサポートされています。
また、(と)も使えます。
使用例
# 残基番号10-50までの炭素のうち、アラニンとシステインに属するものを除く
:10-50&!(:ALA,CYS)
# 水素以外のすべての原子
:*&!H*
:*&!@H=
!@H=5. 距離で指定する
アスパラギン酸残基から5Å以内のナトリウムイオンのように、距離を指定して選択したい場合があります。
このような場合、「指定した残基や原子から指定距離より近い(遠い)原子や分子」のように選択することができます。
距離での選択は「>または’<’」と、「^または:または@」を組み合わせた2文字の演算子(例:<^ や >@ …)と、Å距離を組み合わせて行います。 (^は分子、:は残基、@は原子を表します。)
構文: (アトムマスク) (2文字の距離演算子) (Å距離)
# 10番残基から2.5Å以内の原子
:10<@2.5
# 10から20番残基から2.5Å以内のアラニン残基
(:10-20<:2.5)&:ALA
# アスパラギン酸残基から5Å以内のナトリウムイオン
(:ASP<@5)&:Na+6. よく使う例
| 意味 | コマンド |
|---|---|
| 水素以外のすべての原子 | !@H* |
| タンパク質の全てのバックボーン原子 | @CA,C,O,N,H |
| 水とナトリウム・塩素イオン以外のすべての原子 | !(:WAT,Na+,Cl-) |